Det finns en rad företag som erbjuder genetiska analyser till privatpersoner. För några tusen får du spotta i ett rör, och företaget typar ett antal genetiska markörer, alltså kända platser i genomet som varierar mellan människor. Den informationen, alltså vilka varianter en individ bär på, kan ge lite olika upplysningar. Men det är inte så många genetiska varianter där vi faktiskt vet hur de ska tolkas.
En del information är mest för nöjes skull — saker vi förmodligen redan har märkt eller som vi inte har någon direkt nytta av. De flesta vet vilken ögonfärg de har även utan gentest, till exempel.
Några har kliniskt värde i och med att de speglar variationer i leverns enzymsystem och indikerar förmågan att bryta ner olika läkemedel. CYP2D6, till exempel, är en mycket känd sådan, som då och då typas i vården. Farmakogenetik, kallas den genetik som handlar om variationer i vårt svar på läkemedel, och det är kanske den form av personlig genetik som har störst potential att leverera inom överskådlig tid. Gentester till konsumenter i all ära, men än så länge är de mest en form av dyra leksaker.
Några, slutligen, avspeglar risk för olika sjukdomar. Här är det viktigt att veta, att de flesta sjukdomarna ifråga har en komplex genetisk bakgrund som interagerar med miljöfaktorer. Alltså, det vi hittar är inte en gen som orsakar sjukdomen, utan ett antal genetiska markörer som har ett samband med sjukdomsrisk. (En genetisk markör är en känd variation någonstans i genomet som går att typa; att en markör har ett samband med sjukdomsrisk betyder sannolikt att den ligger i närheten någon variation som orsakar den ökade risken.)
Det mest kända företaget i gentestningbranschen, 23andMe, har varit på tapeten ett par gånger den sista tiden. Den första anledningen var inte så smickrande. Nyligen visade det sig att 23andMe givit felaktiga uppgifter till ett antal kunder för att någon satt ner en platta med prover åt fel håll i en maskin…
Den andra anledningen är desto roligare: nyligen publicerade forskare från 23andMe sin första granskade vetenskapliga artikel: Web-Based, Participant-Driven Studies Yield Novel Genetic Associations for Common Traits publicerades den 24 juni, knappt ett år efter att den skickades in i PLoS Genetics. PLoS, Public Library of Science, publicerar en serie tidskrifter vars innehåll är Creative Commons-licensierat, och alltså kan läsas och spridas vidare av vem som helst (till skillnad från de flesta vetenskapliga tidskrifter, som bara är tillgängliga från universitetsbibliotek och andra instutitioner som betalar multum för prenumerationer).
Det är en artikel som handlar om den första typen av egenskaper ovan: ett antal egenskaper som inte är så medicinskt intressanta, men desto vanligare. Det finns definitivt ett vetenskapligt intresse i att beskriva hur genetiska variationer bidrar till den normala mänskliga variationen i till exempel utseende.
Arbetet är också särskilt intressant på grund av hur datainsamligen gått till. Försökspersonerna är nämligen de av 23andMes kunder som gått med på att att delta med sina data i vetenskapliga försök. De har själva samlat in proverna, genom att skicka sin saliv i ett rör med posten, och information om fenotyper, genom att besvara enkätfrågor på 23andMes hemsida, där de också hämtar sina data. 23andMe har alltså lyckats få försökspersoner att betala för privilegiet att delta i studien!
Den typ genetisk markör som 23andMe jobbar med kallas single nucleotide polymorphism (SNP). Det är ställen där, en enda position i genomet, en bas, skiljer sig mellan individer. Ett A, adenosin, kan till exempel vara utbytt mot cytosin, C. SNP:ar har den goda egenskapen att det finns massor av dem, så det går att typa markörer relativt tätt utspridda över hela genomet och på så sätt beskriva en individs genom i detalj. Genom att kombinera många individers genotypdata med information om dem, går det att statistiskt koppla samman genotyp med fenotyp — alltså att undersöka vilka genetiska varianter som hänger ihop med en viss egenskap. Detta kallas genome wide association (GWAS), kanske på svenska kanske helgenomsassociation. Det är vad 23andMe har gjort.
De har frågat sina kunder vilken hårfärg och ögonfärg de har, ifall de är fräkniga, är morgonmänniskor, har visdomständer, är vänster- eller högerhänta, har glasögon, har tandställning, gillar sötsaker, blir åksjuka och så vidare. Sedan har de formulerat resultaten som 22 antingen-eller: fräknar ja/nej, ögonfärg brun/blå, hårfärg blond/brun, rött hår ja/nej, och så vidare. 22 gånger har de alltså delat upp deltagarna i två grupper: en fall-grupp (”case”) där deltagarna har egenskapen i fråga, och en kontrollgrupp som inte har den. Och så jämförde de, för varje SNP, vilken variant folk har i fallgruppen och vilka de har i kontrollgruppen.
För åtta fenotyper hittade de SNP:ar som statistiskt skiljer sig mellan grupperna, och alltså är associerade med egenskapen — rött hår, fräknar, blont eller brunt hår, bruna eller blå ögon, gröna eller blå ögon, lockigt hår, samt två små konstigheter: tendensen att nysa i vid starkt ljus och förmågan att känna lukten av sparrismetaboliter i urin.
Här ser vi ett så kallat Manhattandiagram för fräknar. Varje punkt är en SNP, och på y-axeln finns p-värdet, som är ett statistiskt mått på hur stor tilltro vi sätter till associationern. Röda punkter är signifikant associerade SNP:ar, alltså de som har p-värden högre än ett visst tröskelvärde.
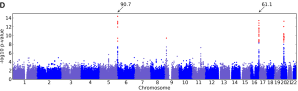
(Bild från artikeln; Eriksson et al. 2010)
Vi lägger märke till att de röda SNP:arna kommer i grupper som bildar små torn i diagrammet. Det är SNP:ar som ligger nära varandra och alltså alla är associerade med egenskapen och den eventuella närliggande underliggande varianten. De associerade SNP:arna leder sedan fram till kandidatgener, alltså troliga misstänkta gener i närheten av markören. Observera uttrycken ”kandidat” och ”misstänkta”… Det är inte alls självklart att genetiska kartläggningar träffar rätt. Kandidatgenerna behöver studeras närmare för att ta reda på vilka varianter det finns i dem, vad de faktiskt gör och hur det går till. Det är förstås också bra ifall resultatet kan upprepas i andra studier. (”The Gay Gene”, en kandidatregion för homosexualitet, är ett känt exempel på en association som inte gått att upprepa — ryktet om den verkar dock ändå inte vilja ta slut.)
Diagrammet ovan gäller fräknar, och här syns tre röda berg — tre regioner där det finns kandidatgener. Två av dem är upprepningar av tidigare associationer och den tredje är ny — det är en SNP som ligger inuti genen BNC2, som därmed är en ny kandidatgen för fräknar. Den liknar BNC1, en bättre känd gen vars proteinprodukt är viktig för hudceller. Det finns alltså en viss logik i att den kunde ha med fräknar att göra. Men som sagt, det återstår att se.
Litteratur
Eriksson N, Macpherson JM, Tung JY, Hon LS, Naughton B, et al. (2010) Web-Based, Participant-Driven Studies Yield Novel Genetic Associations for Common Traits. PLoS Genet 6 e1000993. doi:10.1371/journal.pgen.1000993