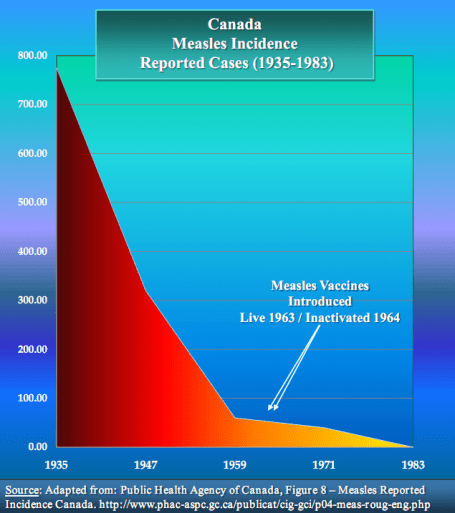
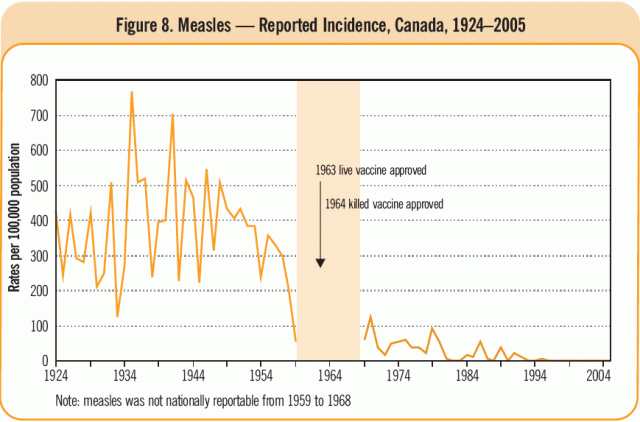
Hört talas om skräp-DNA någon gång? Uttrycket kommer sig av att lejonparten av genomet inte verkar koda för något — varken proteiner eller RNA-molekyler. Det myntades någon gång på sjuttiotalet, men är rätt ute numera. Artiklar om icke-kodande DNA tenderar ha någon mening i inledningen om hur okunniga de var på sjuttiotalet. Det är klart, det klingar kanske lite arrogant att avfärda större delen av genomet som skräp. Men det ligger ändå fortfarande något i det, även om det dyker upp fler icke-kodande sekvenser med kända funktioner.
För det är skillnad på funktion och funktion, och många av de här sekvenserna gör saker som inte är ett dugg konstruktiva för organismen. Vi har mött dem tidigare: transposonerna och (som det heter i Karolinskas hemska översättning) de andra omflyttningsbara DNA-segmenten. De fungerar som en sort parasiter: det enda de kan är att kopiera sig själva och flyttar sig omkring i genomet. Det gör att de kan bli fler och fler på organismens bekostnad. Det vill säga, en del längre varianter, som verkar stamma från retrovirus, har den förmågan — och kodar för omvänt transkriptas. Det finns också kortare varianter som förmodligen uppstått genom att olika oskyldiga RNA-molekyler skrivits om till DNA med hjälp av omvänt transkriptas från en längre retrotransposon.
Vi behöver inte vara medicinska genetiker för att räkna ut att diverse osorterade DNA-sekvenser som sättes in här och där i genomet kan ställa till problem — till exempel genom att störa regleringen av någon viktig gen. Å andra sidan öppnar samma process för nya intressanta mutationer. Organismen gör å sin sida sitt bästa för att hålla de omflyttningsbara elementen i schack. Epigenetisk reglering är, som sagt, en möjlighet.
Det vanligaste parasitiska elementet hos oss människor heter Alu. Det är en av de kortare varianterna, stammar från ett RNA, saknar förmågan att kopiera sig själv, men innehåller en del andra intressanta sekvenser. Den har ett ställe där retinolsyrareceptorn kan binda — vilket gör att en Alu-insättning skulle kunna koppla en gen till vitamin A. Men i det här sammanhanget gäller det en koppling till en annan process: RNA-splitsling (splicing).
Hos oss eukaryoter ligger de kodande delarna av generna utspridda lite här och där. De kodande bitarna kallas exoner, och de icke-kodande bitarna emellan introner. När en gen ska uttryckas skrivs den först om till ett långt RNA med både introner och exoner. Sedan klipps intronerna bort och exonerna sätts ihop till det ett fullständigt mRNA. Det är alltså splitsningen, och den styrs av signalsekvenser.
Men, mycket riktigt, Alu innehåller sekvenser som liknar splitsningssignalerna! Det betyder att Alu-bitar, men några små förändringar, kan leta sig in i mRNA. Vi återkommer till vad de skulle kunna göra där. Först: artikeln — Widespread establishment and regulatory impact of Alu exons in human genes (det är en open access-artikel, så det är bara att hugga in) av Shihao Shen m. fl., som kom ut i Proceedings of the National Academy of Sciences of the USA i februari.
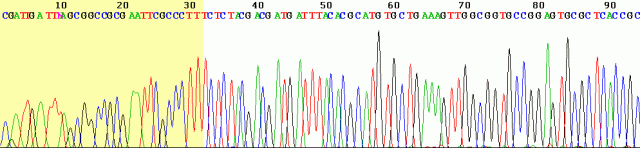
Det är ett till att börja med ett sekvenseringsbaserat arbete, men det är inte DNA-sekvensering utan RNA-sekvensering som gäller. Det betyder alltså att använda massivt parallell sekvensering på cDNA istället för genom-DNA. Det är dels ett sätt att mäta genuttryck — genom att bara räkna hur många gånger en viss mRNA-sekvens dyker upp. Men det är också att sätt att titta på RNA-molekylernas sammansättning, alltså ett utmärkt sätt att se ifall Alu-sekvenser förekommer i mRNA eller inte.
Med en genomdatabas hjälp ställde de sig frågan: Om kända Alu-sekvenser splitsades in, hur skulle skarven mellan Alu och resten av mRNA:t se ut — och sedan letade de efter de skarvarna i ett par publicerade samlingar RNA-sekvenseringsdata från människa.
De tittade efter total 627 Alu-exoner, varav 287 förekom i proverna mRNA. Det är alltså 627 Alu-sekvenser i gener som någon har sett förut, men bara 287 som användes i den här vävnaden — lillhjärnan, cerebellum. Det illustrerar att även för en så väl beskriven organism som människan, där det finns en bra referenssekvens och massor av information om vilka sekvenser som uttrycks, har den samlade biologiska vetenskapen ganska dålig koll på vad som försiggår.
(Förresten, om vi skulle välja en annan teknik som vi behandlat tidigare till att kontrollera resultaten av sökningen i RNA-sekvenser? Rätt gissat, RT-PCR, vilket är precis vad Shen & co också gjorde.)
Nåväl. Det sitter alltså Alu-sekvenser i ett gäng mänskliga gener. Vad är det med det? Jo, en ny bit mRNA, särskilt i början av genen, kan ändra hur mycket protein som produceras från RNA. Att mäta mRNA är en sak, men att få reda på hur mycket protein som produceras är lite knivigare. Det Shen & co gjorde var en teknik med cellodling och reportergener. En reportergen är en gen som är lätt att detektera — det brukar vara ett protein som fluorescererar eller på något annat sätt ger lysande eller färgade celler.
Reportergenen kopplas ihop med den reglerande sekvensen som ska testas. Mängden protein från reportergenen kan mätas som ljus från cellerna och det återspeglar den reglerande sekvensens effektivitet. Nåväl, det fina med det här är att det går att testa olika varianter av sekvensen, genom att införa olika ändringar. Av 15 gener som de prövade var det 10 där Alu-sekvensen verkade göra någon skillnad. En Alu-sekvens i början av mRNA kan alltså göra skillnad. Lite experimenterande med extra stoppkodoner tyder dessutom på att de gör det genom att skapa extra läsramar som börjar i Alu-exonen och tävlar med den kodande genens läsram.
De la också märke till att påfallande många av generna med Alu-exoner tillhörde ZNF-familjen, en serie transkriptionsfaktorer, gener som i sin tur reglerar uttrycket av andra gener. Den här familjen innehåller ett gäng gener som är specifika för primater och som har uttryck som skiljer sig mellan schimpanser och människor. Det är inte direkt några vattentäta bevis, men åtminstone en suggestiv antydan att de där små bitarna av eländigt skräp skulle ha en del i skillanden mellan oss och andra stora apor.
Hur som helst, det här är ett exempel på vad återanvänt genetiskt material kan åstadkomma. Det händer betydligt större — och konstigare saker — under evolutionens gång än enstaka ändrade baser.
Litteratur
Shen S, Lin L, Cal JJ, Jiang P, Kenkel EJ, Stroik MR, Sato S, Davidson BL, Xing Y. (2011) Widespread establishment and regulatory impact of Alu exons in human genes. PNAS 108 ss. 2837-2842