Andrew Gelman sometimes writes that in genetics it might make sense to have a null hypothesis of zero effect, but in social science nothing is ever exactly zero (and interactions abound). I wonder whether that is actually true even for genetics. Think about pleiotropy. Be it universal or modular, I think the evidence still points in the direction that we should expect any genetic variant to affect lots of traits, albeit with often very small effects. And think of gene expression where genes always show lots of correlation structure: do we expect transcripts from the same cells to ever be independent of each other? It doesn’t seem to me that the null can be strictly true here. Most of these differences have to be too small for us to practically be able to model them, though — and maybe the small effects are so far below the detection limit that we can pretend that they could be zero. (Note: not trying to criticise anybody’s statistical method or view of effect sizes here, just thinking aloud about the ”no true null effect” argument.)
Kategoriarkiv: genetik
Inför NBIA25: snäckors och salamandrars kloroplaster
Dagens organell: kloroplasten, stället i växtceller och gröna alger där fotosyntesen äger rum. Fotosyntes i eukaryoter är en fråga om symbios. Det började med någon kusin till cyanobakteriernas förmoder som på något sätt hamnade inuti en annan cell och utvecklades till vad som idag är en organell med visst oberoende i form av egen arvsmassa och egen delning. Kloroplasten producerar energirika organiska molekyler från ljus och kärnan i processen är lite lik en bakvänd version de försurade vakuolerna jag bloggade om igår. Energin från ljus konverteras till en pH-skillnad över ett membran inne i kloroplasten. När vätejonerna sedan passerar ut igen tar cellen vara på energin till energirika bindningar som i ATP och NADPH.
Hur bekvämt vore det inte att ha egna kloroplaster och kunna utvinna sin egen energi från solljus? Om växterna och algerna tagit upp dem så borde väl djur kunna? Det finns minsann ett helt gäng djur som lever åtminstone delvis på fotosyntes: svampdjur, nässeldjur, plattmaskar, blötdjur och sjöpungar (Venn, Loram & Douglas 2008). Men jag tänkte skriva om två extra roliga exempel: en snäcka som lagrar kloroplaster men inte verkar använda dem och en salamander vars ägg lever i symbios med fotosyntetiserande alger.
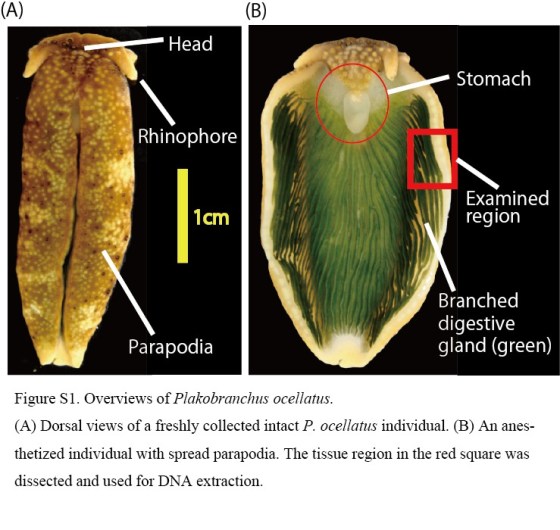
(Figur S1 från Maeda & al 2012. cc:by-3.0)
Det finns snäckor som tar upp kloroplaster från algerna de äter. Fyra snäckelarter har den egenheten att kloroplasterna finns kvar i deras matsmältningsapparat i månader och kallas mycket målande för kleptoplaster. Men hjälper kleptoplasterna verkligen snäckan att producera energi, eller ligger de bara där? Tyvärr verkar det, enligt Christa m.fl. (2013), Plastid-bearing sea slugs fix CO2 in the light but do not require photosynthesis to survive, och Meada m.fl (2012) Algivore or Phototroph? Plakobranchus ocellatus (Gastropoda) Continuously Acquires Kleptoplasts and Nutrition from Multiple Algal Species in Nature, som att de bara ligger där. Snäckorna verkar äta kleptoplasterna, inte använda dem som solceller.
Det är alltid roligt att stöta på en vetenskaplig debatt som en inte visste om: Snäckorna kan tydligen leva väldigt länge utan mat, och den gängse hypotesen har varit att det de får sin näring från de stulna kloroplasternas fotosyntes. Men Christa & co gjorde ett experiment där de höll snäckor i mörker och behandlade dem med en kemikalie som hämmar fotosyntes, och de klarade sig lika bra ändå. Snäckor utan någon möjlighet att få näring från fotosyntes klarade sig lika bra som kontrollsnäckorna i ljus. Åtminstone snäckarterna Elysia timida och Plakobranchus ocellatus verkar inte ha någon nytta av att deras internaliserade kloroplasters fotosyntes. Meada & co tittade på P. ocellatus i sin naturliga miljö, där de främst verkar leva just på att äta alger.
Den fotosyntetiska salamandern, däremot, verkar verkligen ha nytta av sin algpartner. Men det fungerar på ett helt annat sätt. Folk har studerat den länge, men Kerney m.fl (2011), Intracellular invasion of green algae in a salamander host, är ett exempel. Salamandern Ambystoma maculatum bor större delen av sitt liv under jorden, men den lägger ägg i våtmarker. På embryostadiet omges salamandern av gröna alger. I experiment med ljus och mörker klarar sig salamanderägg med alger och ljus bättre än de utan, och vice versa: det verkar alltså vara ett ömsesidigt utbyte mellan salamanderembryon och alger. Det Kerney & co gjorde i den här studien är bland annat att sekvensera algernas rRNA-gener för att placera vilka sorts görna alger det är, titta med elektronmikroskop och hitta alger inuti cytoplasman i salamanderceller, nära mitokondrierna. Trots det såg salamandercellerna normala ut. Det är kanske det konstigaste och intressantaste med hela affären: djur har ett synnerligen komplicerat och aggressivt immunförsvar som är specialiserat på att skilja egna celler från främmande organismer. Hur gör salamandern för att släppa in sin algpartner?
Litteratur
Christa G, Zimorski V, Woehle C, Tielens AG, Wägele H, Martin WF, Gould SB. (2013) Plastid-bearing sea slugs fix CO2 in the light but do not require photosynthesis to survive. Proc Biol Sci 281 doi:10.1098/rspb.2013.2493
Kerney R, Kim E, Hangarter RP, Heiss AA, Bishop CD, Halla BK. (2011) Intracellular invasion of green algae in a salamander host. PNAS 108 doi:10.1073/pnas.1018259108
Venn AA, Loram JE, Douglas AE. (2008) Photosynthetic symbioses in animals. J. Exp. Bot. 29 doi:10.1093/jxb/erm328
Maeda T, Hirose E, Chikaraishi Y, Kawato M, Takishita K, et al. (2012) Algivore or Phototroph? Plakobranchus ocellatus (Gastropoda) Continuously Acquires Kleptoplasts and Nutrition from Multiple Algal Species in Nature. PLoS ONE e42024. doi:10.1371/journal.pone.0042024
Att komma i stämning inför NBIA25: vakuoler och petunians färger
En del av en doktorandtjänst är att undervisa, i mitt fall ungefär en femtedel av min tid. Det mesta av den tiden består av laborationer. Det är å ena sidan kul, för jag gillar laborativt arbete och tror att praktiska övningar är bra för lärandet. Sedan är det väl ingen som verkligen lär sig labbarbete på kurserna, men en får pröva på lite i alla fall. Å andra sidan består arbetsuppgifterna till stor del av att plocka fram och ställa undan prylar. Nu är det dags för årets kurs i Cellbiologi, inte längre den första men fortfarande en av de första kurserna på biologiprogrammet. Cellbiologi kan vara rätt tekniskt — många lådor och pilar! — men laborationerna handlar mycket om roliga observationer en kan göra med rätt enkla medel: mikroskop, hyfsat vanliga kemikalier, grönsaker, jäst och blodceller.
Kursen började den här veckan, men laborationerna kommer inte igång riktigt än. För att komma i stämning tänkte jag läsa och blogga lite kort om roliga artiklar som har (vaga) kopplingar till kursmaterialet. Jag visste till exempel inte att petuniablommor har olika färg beroende på vilket pH de har i sina vakuoler. Vi tar det från början: Vakuoler är en sorts organeller, membranklädda avdelningar inne i celler. Vakuoler kan lagra vatten, salter, näring innehålla enzymer och — i många färgade celler — färgämnen. I petuniablommans fall: anthocyaniner — organiska färgämnen som är pH-känsliga. Det är därför rödkålssaft fungerar som pH-indikator. Jag har skrivit förut om andra genetiska varianter som kan reglera hur mycket anthocyanin som producera i en annan blomma. Att vakuolen är avskärmad från resten av cellen med ett membran betyder att den kan ha en annan kemisk miljö än cytosolen. Petuniablommans vakuoler är surare än resten av cellen och dess pH dras ner av proteiner som sitter i membranet och pumpar vätejoner från cytosolen till vakuolens insida. Blommorna (Petunia x hybrida) är oftast röda eller lila, men det finns genetiska varianter som ger mindre surt pH i vakuolerna och blå blommor.
Det finns åtminstone sju olika gener som kan mutera och orsaka blå blommor. Artikeln ifråga, Faraco m fl (2013). Hyperacidification of Vacuoles by the Combined Action of Two Different P-ATPases in the Tonoplast Determines Flower Color, handlar om hur författarna identifierade en av generna, PH1, som visade sig koda för en en del av ett sådant membranprotein. Tillsammans med produkten av genen PH5 bildar de en enhet som förbrukar energi från ATP till att försura vakuolens insida. Artikeln är fritt tillgänglig och har flera snygga figurer.
(Petunia. Foto: Zirguezi via Wikipedia.)
Litteratur
Faraco m fl (2014). Hyperacidification of Vacuoles by the Combined Action of Two Different P-ATPases in the Tonoplast Determines Flower Color Cell Reports 6 doi:10.1016/j.celrep.2013.12.009
Morning coffee: epigenetic inheritance of odour sensitivity
A while ago I wrote a bit about the recent paper on epigenetic inheritance of acetophenone sensitivity and odorant receptor expression. I spent most of the post talking about potential problems, but actually I’m not that negative. There is quite a literature building up about these transgenerational effects, that is quite inspiring if a little overhyped. I for one do not think epigenetic inheritance is particularly outrageous or disrupting to genetics and evolution as we know it. Take this paper: even if it means inheritance of an acquired trait, it is probably not very stable over the generations, and it is nothing like a general Lamarckian transmission mechanism that can work for any trait. It is probably very specific for odourant receptors. It might allow for genetic assimilation of fear of odours though, which would be cool, but probably not at all easy to demonstrate. But no-one knows how it works, if it does — there are even multiple unknown steps. How does fear conditioning translate to DNA methylation differences sperm that translates to olfactory receptor expression in the brain of the offspring?
A while after the transgenerational effects paper I saw this one in PNAS: Rare event of histone demethylation can initiate singular gene expression of olfactory receptors (Tan, Song & Xie 2013). I had no idea olfactory receptor expression was that fascinating! (As is often the case when you scratch the surface of another problem in biology, there turns out to be interesting stuff there …) Mice have lots and lots of odorant receptor genes, but each olfactory neuron only expresses one of them. Apparently the expression is regulated by histone 3 lycine 9 methylation. The genes start out methylated and suppressed, but once one of them is expressed it will keep all other down by downregulating a histone demethylase. This is a modeling paper that shows that if random demethylation happens slowly enough and the feedback to shut down further demethylation is fast enough, these steps are sufficient to explain the specificity of expression. There are some connections between histone methylation and DNA methylation: it seems that DNA methylation binds proteins that bring histone methylases to the gene (review Cedar & Bergman 2009). Dias & Ressler saw hypomethylation near the olfactory receptor gene in question, Olfr151. Maybe that difference, if it survives through to the developing brain of the offspring, can make demethylation of the locus more likely and give Olfr151 a head start in the race to become the first expressed receptor gene.
Literature
Brian G Dias & Kerry J Ressler (2013) Parental olfactory experience influences behavior and neural structure in subsequent generations Nature neuroscience doi:10.1038/nn.3594
Longzhi Tan, Chenghang Zong, X. Sunney Xie (2013) Rare event of histone demethylation can initiate singular gene expression of olfactory receptors. PNAS 10.1073/pnas.1321511111
Howard Cedar, Yehudit Bergman (2009) Linking DNA methylation and histone modification: patterns and paradigms. Nature reviews genetics doi:10.1038/nrg2540
Journal club of one: ”Parental olfactory experience influences behavior and neural structure in subsequent generations”
Okay, neither chickens nor genetics, really, but a little epigenetic inheritance. Dias & Ressler in Nature neuroscience:
When an odor (acetophenone) that activates a known odorant receptor (Olfr151) was used to condition F0 mice, the behavioral sensitivity of the F1 and F2 generations to acetophenone was complemented by an enhanced neuroanatomical representation of the Olfr151 pathway.
Meaning that the offspring of conditioned mice score higher in an odour potentiated startle test (more about that below), avoid the odour at a lower concentration in an aversion test and have more neurons expressing that odorant receptor in their olfactory epithelium and bulb, counted by betagalactosidase staining in transgenic mice expressing M71, the product of Olfr151, coupled to LacZ.
Furthermore,
Bisulfite sequencing of sperm DNA from conditioned F0 males and F1 naive offspring revealed CpG hypomethylation in the Olfr151 gene. In addition, in vitro fertilization, F2 inheritance and cross-fostering revealed that these transgenerational effects are inherited via parental gametes.
That is, they detect a difference in methylation in one CpG dinucleotide in the 3′ region of the gene.
Comments:
First, I love how the journal does exactly the thing I like to see with figures: below each figure is a link that leads to a data file with the underlying data!
Olfactory behaviour is not my thing, so the tests are new to me, but I’m a bit puzzled by the way they calculate the results from the odour potentiated startle tests. The point is to test whether the presence of the odour make the mice react stronger to a noise. After buzzing the sound 15 times without odour, they perform ten trials with odour plus sound and ten trials with sound only. But in calculating the score, they use only the difference between the first trial with odour and the last trial with sound only divided by how much the mouse reacted to the last of the first 15 sounds. Maybe this is standard, but why throw away the trials in between?
It is not only the olfactory potentiated startle and the sensitivity test, but the staining results. Again, this is not my area, but the results all seem to point to increased sensitivity in the offspring of the treated animals. They react stronger in the startle test, react at lower concentration in the avoidance test and they (in this case, the transgenic mice) have more neurons expressing M71. The cross fostering and the fact that the males were treated but not the females points to genuine inheritance. So, how does the treatment get into the germline? It has to cross that boundary and enter the sperm somehow. Unless there is some mysterious way for information from the central nervous system to travel to the testis, acetophenone must affect the spermatogenesis as well as the olfactory neurons.
All this is very hypothetical, so a little skepticism is not surprising. Gonzalo Otazu wrote in a comment on the Nature news webpage:
The statistical tests in the paper, both for the behavioral measurements as well as for the size of the M71 glomeruli , use as n, number of samples, the number of F1 and F2 individuals. This would be fine if the individuals were actually independent samples. However, they arise from a presumably small number of FO males. The numbers of FO males are not given in the paper. This is a major concern given that there is a lot of variability in the levels of expression of olfactory receptors in these mice that might be inheritable …
I think this is a good point but it will not be solved, as the comment later suggests, by adjusting the degrees of freedom of the test. From the F1 generation and on, genetic differences between the treatment groups, if they do exist, will amplify into a bias issue. That is, it is a systematic difference that might be bigger or smaller than the treatment effect and go in the same or opposite direction — we don’t know. However, the bias should not be there all the time, and not in the same direction, so it strengthens the authors’ case that they’ve done the treatment at least twice (with C57B/6J and with M17-LacZ mice, if not more times).
Maybe my preference for genetics is showing, but I feel the big unadressed alternative hypothesis in most transgenerational effects experiments is cryptic heritability. If you divide individuals into two groups, treat one of them and look for treatment effects in the offspring, you need to be sure that there are not genetc differences between the founders of the two groups. In the subsequent generations, genetic and non-genetic inheritance will be counfounded by design.
Again, randomisation and replication will help, but to be really sure, maybe one can use founders of known relatedness to create a mixed population — say take founders from full-sibships and split them equally between treatment groups, allowing segregation to randomise the genotypes of the next generation. It doesn’t say in the methods — the authors might even have done something like this. One could even use a genetic mixed model that includes relatedness as to estimate treatment effects over in the prescence of a genetic effect. I have a suspicion this experiment would require a much larger sample size, which means more time, work and animals — but I also believe that many would find confounding genetic variation more plausible than transgenerational epigenetic effects of unknown mechanism.
Literature
Brian G Dias & Kerry J Ressler (2013) Parental olfactory experience influences behavior and neural structure in subsequent generations Nature neuroscience doi:10.1038/nn.3594
Journal club of one: ”Short copy number variations potentially associated with tonic immobility response in newly hatched chicks”
(‘Journal club of one’ will be quick notes on papers, probably mostly about my favourite topics — genetics and the noble chicken.)
Abe, Nagao & Inoue-Murayama (2013), recently published this paper in PLOS ONE about copy number variants and tonic immobility in two kinds of domestic chicken. This obviously interests me for several reasons: I’m working on the genetic basis of some traits in the chicken; tonic immobility is a fun and strange behaviour — how it works and if it has any adaptive importance is pretty much unknown, but it is a classic from the chicken literature — and the authors use QTL regions derived directly from the F2 generation of cross that I’m working on — we’ve published one paper so far on the F8 generation.
Results: They use arrays and qPCR to search for copy number variants in three regions on chromosome one in two breeds (White Leghorn and Nagoya, a Japanese breed). After quite a bit of filtering they end up with a few variants that differ between the breeds. The breeds also differ in their tonic immobility behaviour with Leghorns going into tonic immobility after three attempts on average and lying still for 75 s and Nagoya taking 4.5 attempts and lying for 100 s on average. But the copy number variants were not associated with tonic immobility attempts or duration within breeds, so there is not really any evidence that they affect tonic immobility behaviour.
Comments:
Apart from the issue that the regions (more than 60 Mb) will contain lots of other variants, we do not know whether these regions affect tonic immobility behaviour in these breeds in the first place. The intercross that the QTL come from is a wild by domestic Red Junglefowl x White Leghorn cross, and while Nagoya seem a very interesting breed that is distant from White Leghorn they are not junglefowl. When it comes to the Leghorn side of the experiments, I wouldn’t be surprised White Leghorn bred on a Swedish research institute and a Japanese research institute differed quite a bit. The breed differences in tonic immobility is not necessarily due to the genetic variants identified in this particular cross, especially since behaviour is probably very polygenic, and an F2 QTL study by necessity only scratches the surface.
In the discussion the authors bring up power: There were 71 Nagoya and 39 White Leghorn individuals and the experiment might be unable to reliably detect associations within the breeds. That does seem likely, but making a good informed guess about the expected effect is not so easy. A hint could come from looking at the effect sizes in the QTL study, but there is no guarantee that genetic background will not affect them. I don’t know really what this calculation comes from: ”Sample sizes would need to be increased more than 20-fold over the current study design” — maybe 11 tested copy number variants times two breeds? To me, that seems both overly optimistic, because it assumes that the entire breed difference would be due to these three QTL on chromosome 1, and overly pessimistic, since it assumes that the three QTL would fractionate into 11 variants.
Finally, with all diversity in the chicken, there’s certainly a place both for within and between population studies of various chickens with all kinds of genomic! Comparing breeds with different selection histories should be very interesting for distinguishing early ‘domestication QTL’ from ‘productivity QTL’ selected under modern chicken breeding. And I wish somebody would figure out a little more about how tonic immobility works.
Literature
Abe H, Nagao K, Inoue-Murayama M (2013) Short Copy Number Variations Potentially Associated with Tonic Immobility Responses in Newly Hatched Chicks. PLoS ONE 8(11): e80205. doi:10.1371/journal.pone.0080205
Tre rekommendationer
1. Imre Lakatos, Science and pseudoscience
Vetenskapsfilosofi är något folk i allmänhet, och jag själv i synnerhet, borde läsa mer av och tänka mer på. Svårt verkar det också — trots käcka bilder och Richard Feynman-citat. Imre Lakatos var en vetenskapsfilosof vars namn jag stött på här och där, men inte läst något av förrän alldeles nyligen. Via Dynamic Ecology hittade jag en länk till ett av hans föredrag:
As opposed to Popper the methodology of scientific research programmes does not offer instant rationality. One must treat budding programmes leniently: programmes may take decades before they get off the ground and become empirically progressive. Criticism is not a Popperian quick kill, by refutation. Important criticism is always constructive: there is no refutation without a better theory. Kuhn is wrong in thinking that scientific revolutions are sudden, irrational changes in vision. … On close inspection both Popperian crucial experiments and Kuhnian revolutions turn out to be myths: what normally happens is that progressive research programmes replace degenerating ones.
2. Trudy Mackay, The genetic architecture of quantitative traits
Jag var på ESEB-konferensen i Lissabon i somras och det var helt fantastiskt på snart sagt alla sätt (mitt glada jollrande om konferensen finns här och här). Bland det bästa var det här föredraget från Trudy Mackay — en av dem som bokstavligen skrev boken om kvantitativ genetik. Mackay pratade om hennes grupps arbete med genetisk kartläggning av kvantitativa egenskaper i bananflugor och om gen–gen-interaktioner, som det verkar finnas förvånansvärt många av — vilket gör saker mer komplicerade och roligare. Naturligtvis blir det ganska tekniskt efter hand och presentationsbilderna är tyvärr oläsliga, men det rekommenderas ändå varmt. Dessutom börjar Mackay med en genomgång av vad kvantitativ genetik är, varför det är viktigt och varför en kan lära sig så mycket från bananflugor. Det här är ett av mina favoritcitat:
Those of you who don’t work with flies might be surprised to learn that flies vary for any phenotype that an investigator has the imagination to develop a quantitative assay for.
Några fler föredrag från konferensen har publicerats och det kommer bli fler på ESEB2013:s YouTube-kanal.
3. Richard Lenski, Telliamed Revisited
Richard Lenski är evolutionsbiolog som förmodligen är mest känd för sitt Long Term Evolution Experiment — ett evolutionsexperiment med bakterien Escherichia coli. På sin blogg skriver han bland annat om vad de hittat i det experimentet — börja till exempel med den här posten:
Fitness is the central phenotype in evolutionary theory; it integrates and encapsulates the effects of all mutations and their resulting phenotypic changes on reproductive success. Fitness depends, of course, on the environment, and here we measure fitness in the same medium and other conditions as used in the LTEE. We estimate the mean fitness of a sample from a particular population at a particular generation by competing the sample against the ancestral strain, and we distinguish them based on a neutral genetic marker. Prior to the competition, both competitors have been stored in a deep freezer, then revived, and acclimated separately for several generations before they are mixed to start the assay proper. Fitness is calculated as the ratio of their realized growth rates as the ancestor and its descendants compete head-to-head under the conditions that prevailed for 500 … or 5000 … or 50,000 generations.
Pig and chimp dna just won’t splice — eller: ta en titt på Wikipedia och mejla mig
Okej, titeln hänvisar till South Park som ju verkligen inte är en särskilt bra serie. Men det visar sig att South Park-figuren Dr. Alphonso Mephesto (så!) har en sorts verklig motsvarighet — verkligheten överträffar dikten och så vidare. Det finns tydligen en genetiker som driver en egen hypotes om människans utveckling: att vi kommer från hybrider mellan schimpanser och grisar. Det finns som bekant ingen åsikt som är så konstig att det inte är någon som driver den. Och på något sätt har The Daily Mail, som är tämligen ökänd för sin dåliga vetenskapsjournalistik, fått för sig att skriva om det. Och direkt från the Daily Mails hemsida kommer den till Aftonbladet.
Eugene McCarthy heter han som driver idén att människan är en schimpans–grishybrid. Det är han förmodligen ganska ensam om att tro — varför framgår nog om en läser hans väldigt långdragna hemsida. Jag har bara läst den länkade artikeln där han än så länge kommit fram till att det inte finns något som tyder på att schimpanser och grisar kan få fertil avkomma, att det finns andra förklaringar till kroppsliga likheter mellan människor och grisar samt att människan genetiskt inte särskilt liknar grisen men däremot schimpansen. Vad som talar för hans hypotes har han inte hunnit med.
Men det var inte hypotesen som sådan jag tänkte skriva om utan vetenskapsjournalistik. (Om du verkligen undrar om grisarna och schimpanserna, se PZ Myers bloggpost. Myers är för övrigt professor i biologi, så hans akademiska position smäller klart högre än min, och han har valt ett annat South Park-klipp som illustration.) Det är såklart väldigt lätt att skälla på de som skrivit artiklarna i Daily Mail och Aftonbladet — det borde väl vem som helst fatta att ”teorin” ifråga är ett påhitt från en tok? Eller? Jag vet faktiskt inte! Jag tycker kanske att den som skriver ett referat borde kunna ta sig tillbaka till källan och se att det inte är något publicerat forskningsresultat, utan en ganska suspekt egen hemsida. Men när det gäller påståendet att människan är en schimpans-grishybrid? Det låter absurt, men det finns det också en hel del riktig forskning som gör. Forskare och vetenskapskommunikatörer är dessutom väldigt förtjusta i att hitta på slående och uppseendeväckande rubriker och sammanfattningar. Det kanske faktiskt inte är så lätt att veta vad som är rimligt och inte.
Det är utan tvivel så att forskare — inklusive små doktorander som undertecknad — ibland kan vara dryga på Twitter eller skriva arga mejl när de tycker någon har publicerat något dumt. Och de är, precis som journalister och reportrar upptagna och under ständig tidspress. Men de flesta bör vara vana vid och intresserade av att förklara vetenskap. Så om du är reporter, sitter med en artikel i knät och undrar vad det betyder och om det är trovärdigt — ring någon! Skicka ett mejl! Det ingår faktiskt i vårt jobb att dela med oss av kunskap. Även om någon som jag absolut inte är expert på det mesta inom biologi, så har vi i alla fall haft mycket övning i att läsa och utvärdera vetenskapliga påståenden.
Isfiskarnas färglösa blod och förlorade hemoglobingener
Blodet är rött på grund av det syrebärande proteinet hemoglobin. Nu finns det i och för sig många andra djur som har andra lösningar, men bland ryggradsdjur är det rött blod och hemoglobin som gäller. Vi klarar oss inte något vidare utan — det finns flera olika sorters genetiska anemier som beror på mutationer i generna för hemoglobin. Men det finns fiskar kring Antarktis, isfiskar, som klarar sig utan både hemoglobin och röda blodkroppar. Det är inte några avlägsna släktingar som skildes från andra fiskar innan hemoglobinet kom till — de kommer från förfäder som hade hemoglobin men har förlorat det.
Sidell & O’Brien (2006) har en bild på isfiskarnas grå blod: se figur 1.
Det finns flera gener som kodar för olika delar av hemoglobinproteinet. Isfiskarna saknar proteinet, saknar helt gener för betaglobin men har kvar rester av en gen för alfaglobin. Kopior av gener som muterat sönder på olika sätt brukar kallas pseudogener. En gen kan bli pseudogen om en bit av den försvinner eller en mutation introducerar en ny signalsekvens. Det kan vara en stoppsignal, så att proteinet slutar för tidigt eller en splitssignal, så att icke-kodande sekvenser som inte brukar höra hemma i den kodande delen hamnar där — och så vidare. Isfiskarnas alfaglobingen är bara ett fragment och en art har dessutom tappat en bit till. När en gen väl förlorat sin funktion finns det inte mycket som hindrar att den muterar sönder ännu mer eller försvinner. Och att försvinna helt verkar vara just det öde som drabbat isfiskarnas betaglobin.
På land fungerar det som sagt väldigt dåligt att vara ryggradsdjur utan hemoglobin. Men syre löser sig bättre i kallt än varmt vatten och dessutom har isfiskarna ovanligt stora hjärtan och ovanligt mycket blod. Åsikterna går isär om ifall det färglösa blodet är en anpassning, alltså något som fungerar bättre än rött blod i kallt vatten, men om vi ska tro Sidells & O’Briens sammanfattning av läget är svaret antagligen nej. Det är uppenbarligen en lösning som fungerar, men om den har någon fördel är det oklart vilken det skulle vara.
(En isfisklarv. Foto: Uwe Kils. CC:BY-SA 3.0 via Wikimedia Commons.)
Men det finns en art hemoglobinlösa isfiskar, Neopagetopsis ionah, som avviker från mönstret (Near, Parker & Dietrich 2006). Den här arten har kvar en sin trasiga betaglobingen, och den har faktiskt inte bara en utan två pseudogener av betaglobin, som verkar komma från olika ursprung och är trasiga på olika sätt. Det verkar som att N. ionah behåller en äldre längre version av sekvensen — efter att betaglobin gått sönder men innan den förlorats helt. Som om det behövdes ett exempel på att genom är röriga och att arters evolutionära historia ibland är något bisarr: pseudogenerna är alltså olika fragment av olika betaglobingener, där den ena mest liknar den från en avlägsnare släkting till isfiskarna. Pseudogenerna verkar ha uppstått i en korsning mellan en anfader eller -moder till isfiskarna och en föregångare till släktingarna i Nothotheniidae där det blivit en misslyckad rekombination mellan de olika versionerna av genen.
Normalt innehåller globinfamiljen både vuxenvarianter och embryovarianter av hemoglobin. Det ovanstående, i alla sin rörighet, är alltså inte ens hela saken. Och isfiskarna saknar röda blodkroppar också, men det är en annan historia.
Litteratur
Near, Parker & Dietrich (2006). A Genomic Fossil Reveals Key Steps in Hemoglobin Loss by the Antarctic Icefishes. Mol Biol Evol doi: 10.1093/molbev/msl071
Sidell & O’Brien (2006). When bad things happen to good fish: the loss of hemoglobin and myoglobin expression in Antarctic icefishes. J Exp Biol doi: 10.1242/jeb.02091
Populär/vetenskapligt föredrag om hönskammar imorgon
Jag har helt missat att göra reklam för detta, men imorgon klockan fyra ska jag hålla ett kort föredrag om hönskammen som en del av Linköpings universitetsbiblioteks Fängslande forskning på femton minuter. Jag kommer använda kammen, som är ett sexuellt ornament hos höns, som exempel för att berätta om hur vi försöker reda ut den genetiska grunden för skillnader mellan tama och vilda höns. Orden ”Redan Charles Darwin …” kommer nämnas. Dessutom miljöteknik, medicinsk teknik och tunnfilmsfysik. Jag utgår ifrån att allt kommer vara roligt, men jag vet att Anette Karlssons forskning om muskler i magnetkamera ensamt skulle varit värt ett besök.