Detta har hänt: Det finns röda och gula gyckelblommor som bor i olika delar av Californien. I ett område där de möts, en så kallad hybridzon, kan de korsa sig, men populationerna är ändå till största del separerade bland annat för att röda och gula blommor pollineras av olika djur som föredrar blommor av respektive färg. Det vore alltså extra kul att veta vilka genetiska varianter som skiljer röda och gula blommor, eftersom de också är varianter som påverkar reproduktiv isolering mellan olika populationer av gyckelblommor. Anthocyaniner är röda och lila färgämnen i växter; gener inblandade i anthocyaninhantering är såklart huvudmisstänkta. Därför gjorde Streisfeld & Rauscher (2009) en genuttrycksundersökning av de gener som kodar för de proteiner som tillverkar anthocyanin i de här blommorna. Mycket riktigt: regleringen av anthocyaninsyntes skiljer sig mellan blommorna, och genetisk kartläggning ledde Streisfeld & Rauscher till två regioner i genomet. I en av dem ligger genen Dfr, som kodar ett av enzymerna i kedjan. Det var en väldigt rimlig hypotes att någon variant i Dfr skulle vara inblandad, men så verkar inte vara fallet. Tillbaka till ritbordet: finns det någon annan gen, nära Dfr, som kan vara inblandad istället? Där står vi när den dagens artikel börjar.
Det är inte meningen att den här bloggen ska handla helt om gyckelblommor, men det är var också för bra att inte skriva om: genetisk kartläggning, genuttryck, naturligt förekommande varianter som påverkar egenskaper. Växter eller djur spelar mindre roll: den här artikeln är ett typiskt exempel på genetisk forskning jag är intresserad av. (Det är i alla fall inte precis samma art som förra gångerna: Mimulus aurantiacus istället för guttatus …) Även om jag är förtjust i diagram och grafer för egen del är jag ganska usel på att titta noga på dem. Därför tänkte jag presentera den här artikeln i form av några av dess figurer. Det är också en anledning att jag föredrar att skriva om open access-artiklar, där det är fritt fram att återanvända bilder. Alla figurerna kommer från Streisfeld, Young & Sobel (2013) och omfattas av Creative Commons Attribution-licensen.
Figur 1 — en karta över hybridzonen och ett par fotografier av blommorna. Gula blommor finns åt öster och de röda åt väster.
Det här är en karikatyr av hur det röda färgämnet anthocyanin tillverkas i en växt: en serie kemiska rekationer som katalyseras av olika enzymer och som regleras av ett gäng transkriptionsfaktorer (delvis oklart vilka de alla är). Flera av generna i den här kedjan uttrycks olika mycket i röda och gula blommor — de reglerande transkriptionfaktorerna är de geometriska formerna till höger, och det är bland dem som den eftersökta genen (antaligen) finns. Det här är vad som på fikonspråk heter trans-reglering. Trans betyder typ ”på andra sidan”, och det betyder i det här sammanhanget att en gen regleras från någonannanstans i genomet, av en annan gen. Motsatsen är cis-reglering: reglering på nära håll. En transreglerande genetisk variant är något som påverkar en gen så att den i sin tur påverkar de andra gener som den reglerar.
Så de gav sig ut på jakt efter den saknade transreglerande genen. Anthocyaninsyntes är rätt väl beskrivet i en del andra växter, så de började med att leta upp potentiella transkriptionsfaktorer. Det finns ingen referenssekvens för Mimulus aurantiacus, så de vände sig mot släktingen M. guttatus och fann tre lovande kandidater som de kunde hitta uttryckta i blommorna.
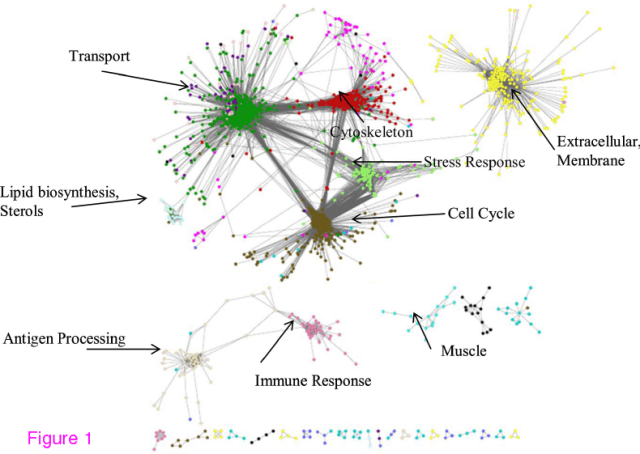
Nästa figur är ett släktträd men inte av arter utan av gener, baserat på hur lika proteinernas aminosyrasekvenser är varandra. Pilarna pekar ut de tre misstänkta transkriptionsfaktorerna MaMyb1, 2 och 3. Generna heter alltså Myb1, 2 och 3 och de två första bokstäverna står för Mimulus aurantiacus. De jämförs med andra gener från andra arter, bland annat några ”Mg” (Mimulus guttatus) och ”At” (Arabidopsis thaliana; backtrav). MaMyb1 och MaMyb2 hamnar på samma gren som andra gener som är kända för att reglera anthocyanin i M. guttatus.
Tre gener alltså… Dags för lite genetisk kartläggning. Den misstänkta genen måste ligga ganska nära Dfr, på fikonspråk: vara länkad till Dfr. Hur avgör en det? Jo, genom att typa genetiska markörer nära MaMyb1, MaMyb2, MaMyb3 och Dfr, och se hur ofta det förekommer rekombinationer emellan dem; rekombinationsfrekvens är ju ett mått på genetiskt avstånd. De tittade i en experimentkorsning mellan röda och gula blommor, och MaMyb1 och 3 var inte länkad till Dfr, men avståndet till MaMyb2 var ungefär 11 cM (vilket är typ 11% rekombinationsfrekvens). Okej, har genotyp på MaMyb2 någon effekt på anthocyanin? Panel A visar effekten av genotyp hos MaMyb2 (svart stapel) och Dfr (vit stapel) i samma korsning — och mycket riktigt, MaMyb2 är associerat med anthocyanin. Dfr är det inte!
Panel B visar samma typ av resultat, men i naturligt förekommande hybrider från hybridzonen ovan. Det finns ett tydligt mönster där en viss genotyp (färg i diagrammet) på markörer i MaMyb2-genen överensstämmer med gul respektive röd färg. Markörer nära Dfr har inget tydligt mönster.
Okej, så om en vill veta ifall en gen utför en viss funktion, i det här fallet gör blommor röda, vad är ett bra experiment? Slå ut genen och se om funktionen också går sönder! Virus-induced gene silencing är en metod som lite liknar genterapi. Växter tolererar naturligtvis ogärna virus, och metoden går ut på att sätta in en kopia av en växtgen i ett virus, så att växten överreagerar och stänger ner uttrycket av den egna genen av bara farten. Nästa bild, panel A, visar ett exempel på resultatet. När MaMyb2 tystas blir en blomma som normalt skulle bli röd (om inte helt så delvis) gul:
Panel B visar att den synliga effekten också är där på molekylär nivå. De svarta visar genuttryck i normala blommor och de vita genuttryck i de virusbehandlade: Inte bara MaMyb2 utan också andra gener i anthocyaninsystemet är nedreglerade — vilket pekar på att MaMyb2 reglerar dem.
Så, MaMyb2 är definitivt inblandad i färg. Om den stängs av har det en effekt både på genuttryck och färg. Panel C — och det är den sista bilden vi ska titta på idag — visar resultatet av ett experiment för att pröva om det är just detta som händer med en naturligt förekommande genetisk variant i de röda och gula blommorna.
Tricket är att med pyrosekvensering (en sekvenseringsteknik som är lite annorlunda den den jag brukar prata om, men den har sin speciella roll) går det faktiskt att mäta genuttryck på allelnivå — i heterozygota individer (blommor som har både allelen för röda och för gula blommor), vilken av allelerna är det som uttrycks mest? De grå staplarna visar andelen av den röda allelen — och den är nära 1. Den gula allelen uttrycks nästan inte alls.
Det här är ett utmärkt tillfälle att introducera en teknisk term att skrämma vänner och bekanta med: detta betyder att MaMyb2 har en så kallad cis-eQTL. QTL står för quantitative trait locus — en plats i genomet (ett locus) som påverkar en kvantitativ egenskap. e:et står för expression — alltså genuttryck. Cis betyder som sagt att varianten är nära. Panel C tyder på att det finns en genetisk variant i närheten som påverkar regleringen av MaMyb2 och i sin tur blommornas färg. Sammantaget ger den här artikeln utmärkt stöd för en (ännu okänd) variant nära MaMyb2 som en av de två generna som förklarar skillnaden i färg mellan gula och röda blommor. Och där slutar vi för idag.
Litteratur
Streisfeld MA, Rausher MD. (2009) Altered trans-Regulatory Control of Gene Expression in Multiple Anthocyanin Genes Contributes to Adaptive Flower Color Evolution in Mimulus aurantiacus. Molecular biology and evolution 26 ss. 433-44 doi: 10.1093/molbev/msn268
Streisfeld MA, Young WN, Sobel JM (2013) Divergent Selection Drives Genetic Differentiation in an R2R3-MYB Transcription Factor That Contributes to Incipient Speciation in Mimulus aurantiacus. PLoS Genet 9 e1003385 doi:10.1371/journal.pgen.1003385